目前全长转录组测序(Iso-seq)技术的优势逐渐被广大学者所熟知,主要体现在对新基因/新isoform的挖掘、对可变剪切(AS)、可变聚腺苷酸化(APA)、融合基因、LncRNA等的预测及分析。可谓是三代测序一出手,基因组注释“全”都有。下面小编以2篇经典案例带大家深入了解全长转录组在揭示转录组复杂性、辅助基因组注释方面的具体应用~
文献名称:全长转录组测序揭示斑马鱼高分辨率基因注释[1]
发表期刊:Genome Research
发表时间:2018 年7 月
样本选择:转录抑制剂处理前后的15个斑马鱼胚胎,处理后的胚胎停留在ZGA(合子基因激活)阶段前。斑马鱼早期胚胎发育由其母本遗传物质驱动,经历ZGA阶段后一些发育必需基因才开始发生转录。
实验设计
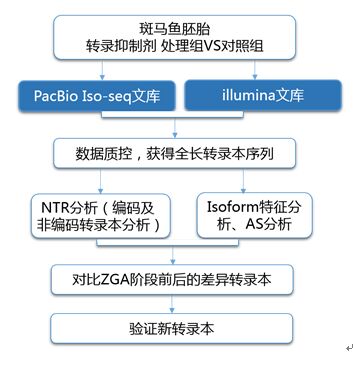
图1 实验设计流程图[1]
实验结果
01 全长转录组数据概览
作者利用三代测序获得斑马鱼胚胎的全长转录本序列信息,并比对参考基因组,共有18,777条转录本成功比对,仅3.6%未能比对上,map率远高于短读长测序的结果(低于80%)。在对新转录本的研究中,获得了4,205条(22.4%)被认为是潜在新转录区(NTR)的转录本与5,295条(28.2%)新isoform。 图2 长读长转录组特征概览[1]
02 NTR区转录本分析
作者对有高度编码潜力的NTR进行序列同源性和功能域分析,共鉴定到3,255个具有高度编码潜能的NTR。首次发现斑马鱼的一个NTR位点与人的HIST2H2BE基因具有同源性,是组蛋白变体的直系同源,且此变体仅在对照组样品中出现,因此猜测此变体起源于斑马鱼的受精卵。
将非编码的NTR进行保守性和功能分析,发现有258个非编码NTR的保守性在进化中有所提高。通过与Rfam数据库比对,共匹配上76条,其中一个NTR与mir-548相匹配,只在处理组样品中表达,推测其RNA多数从母本中来;它的一个3’端较短的isoform则出现在对照组中。匹配上的miRNA位于延长的3’端,可能是假定的靶点或miRNA结构本身,并调控了母本向合子转变的过程。
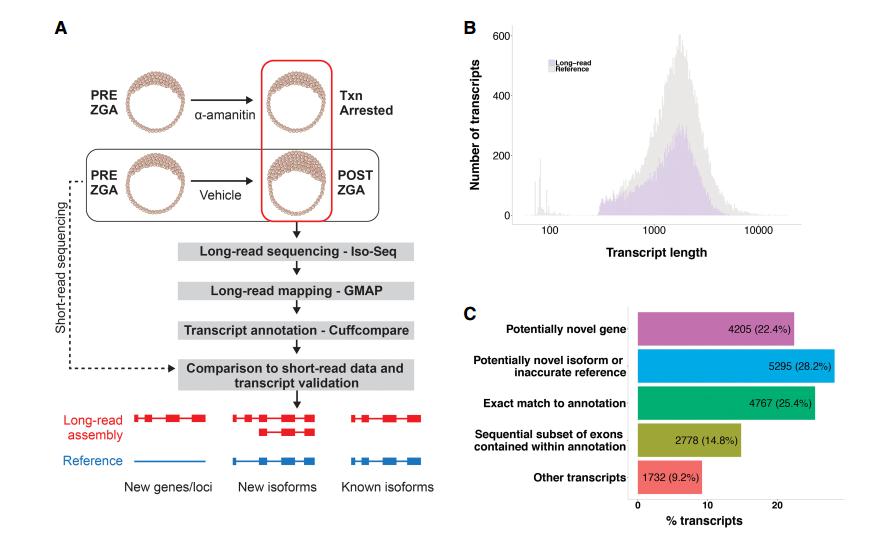
图3 推测的新miRNA同系物[1]
03 新isoform分析
作者对已知基因位点的新isoform进行了分析,新发现的可变剪切事件超过2,000件。利用二代转录组数据将其进行定量,通过对比胚胎早期和晚期样本,发现胚胎晚期阶段可变3’UTR和内含子保留的isoform有明显的增加。对可变剪切事件的验证中,作者还发现一条含有多个mir-430重复序列的8kb新转录本,是胚胎早期发育的重要驱动。该研究揭示了斑马鱼胚胎转录组的复杂性,并为其提供了高分辨率的注释资源。
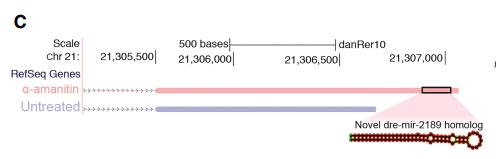
图4 可变剪切分析[1]
文献名称:全长转录组测序揭示自交系玉米遗传多样性[2]
发表期刊:Plant Biotechnol Journal
发表时间:2018 年12 月
样本选择:自交系玉米W64A授粉后16天的硬质胚乳
实验设计
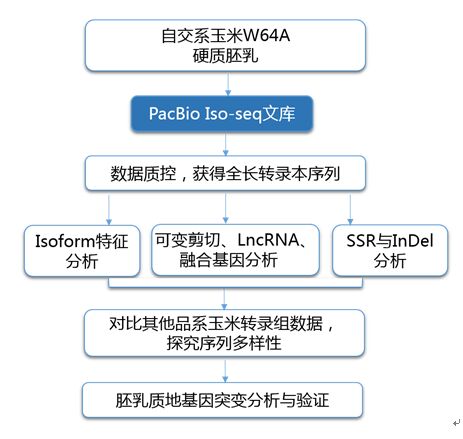
图5 玉米实验设计流程图[2]
主要结果
01 全长转录组测序数据概览
作者利用全长转录组测序共获得166,693条高质量全长转录本,将这些序列比对到参考基因组上,鉴定到3,399个新基因位点和75,711个已知位点的新isoform。对新isoform的分析发现W64A与B73品系具有相似的可变剪切模式,可变3’与可变5’形式比外显子跳跃和内含子保留形式更多。除此之外,作者还对lncRNA和融合基因进行了数量统计和分类比较。经过严格的参数设定,共识别了1,051个SSR和243个InDel,并且分别有63%和58.8%的分子标记得到了验证。
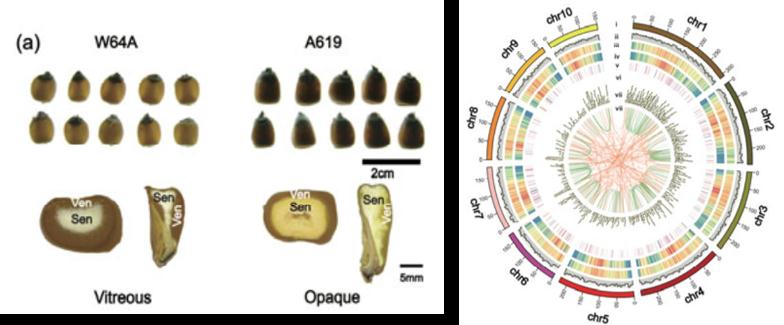
图6 玉米胚乳性状表现/转录本特征分析[2]
02 胚乳质地的相关基因序列多样性分析
为理解透明胚乳形成原因,作者对涉及胚乳质地的相关基因进行了探究。硬质相关基因有相似的拷贝数,包括zein蛋白和non-zein蛋白、酶和转录因子基因等,平均序列多样性为3.92%,最高达26.9%。编码基因中“TTAT”的缺失造成阅读框移码突变从而编码提前终止,使得一个18-kDa的蛋白在W64A品系中不表达,而在B73品系中表达。之后对比这两个品系玉米的其他基因,包括19-和22-kDa蛋白的编码基因在内,有超过20%表现出序列变化。O1基因编码区的2个InDel使O1蛋白的氨基酸数量发生改变,从而产生不透光的胚乳。这些基因的突变导致蛋白编码提前终止,因此对玉米胚乳的性状产生了影响。本项研究中,全长转录组测序不仅揭示了玉米胚乳转录组的复杂性,还为优良性状筛选和遗传育种提供了丰富的分子标记资源。
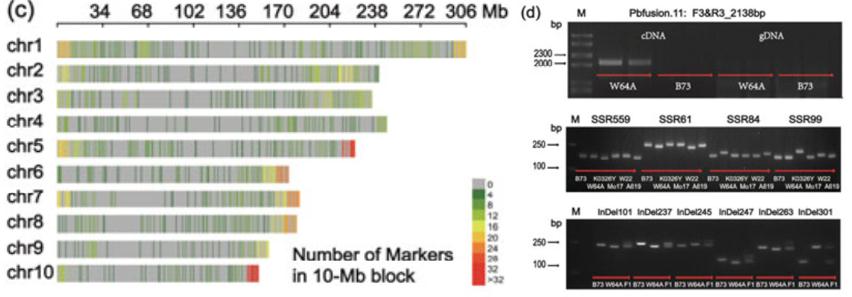
图7 SSR和InDel分布及验证[2]
以上两篇文献借助Iso-seq技术实现了全长cDNA序列从5’到polyA尾的全部信息捕获,有利于对编码区域、调控元件和分子标记的精准识别,极大地提高了对应物种的注释完整度,为后续研究贡献了丰富的高精度注释资源。
作为目前国内三代测序的优质服务商,安诺优达拥有10台PacBio Sequel测序平台,搭建了业界先进的基因组组装、结构变异检测、全长转录组等数据分析体系,积累了丰富的物种及合作项目经验,具备快速的项目交付周期。专业的技术团队致力为您提供优质、高效的测序及分析服务,期待与您的合作。
参考文献:
[1] Nudelman German, Frasca Antonio, Kent Brandon, et al. High resolution annotation of zebrafish transcriptome usinglong-read sequencing[J]. Genome Research, 28, 1415-1425.
[2] Zhou Yong, Zhao Zhixuan, Zhang Zhiyong, et al. Isoform sequencing provides insight into natural genetic diversity in maize[J]. Plant Biotechnol Journal.
邀您关注近期会议
2019年5月17日安诺优达生命科学研究院联合上海交通大学共同举办“表观遗传学与前沿基因组学技术研讨会”。会议以“单细胞和三维基因组学技术”为探讨对象,拟邀多位院士、专家、科研工作者分享两大技术在相关研究领域的最新前沿动态、研究成果。期待您的参与~


