随着2018国自然“大选”初步落下帷幕,各个研究领域的火热程度也以中标项目的形式呈现在大众眼前。在小编感兴趣的表观遗传研究领域,RNA甲基化的研究成为了今年表观中标高产地,RNA m6A也成了表观领域的研究热词。但是你是否知道,6mA是什么?其研究现状如何?本文将给你详细的解答!
DNA 6mA是什么?
真核生物中常见的甲基化类型是胞嘧啶5位碳原子的甲基化,即5mC,其实生物体内还存在着其他甲基化类型。N6-甲基腺嘌呤(N6-methyladenine,6mA)是指腺嘌呤6位氮原子的甲基化修饰,近年来发现在真核生物DNA表观修饰中起重要作用。

DNA 6mA与RNA m6A的联系与区别
二者都是碱基序列上腺嘌呤的6位氮原子甲基化修饰,但研究层面不同。在RNA层面,m6A是RNA最主要的甲基化修饰形式,在哺乳动物中,m6A修饰的比例为0.1-0.4%,平均每条mRNA有3-5个m6A位点;而在DNA层面,其表达丰度非常低,在哺乳动物中,平均每百万个腺嘌呤中只有不到10个6mA位点。目前已研究的物种6mA丰度如下表所示[1](计算单位p. p. m (part permillion dAs))。

二者在动态变化过程中涉及的甲基化/去甲基化酶也不同[2]:
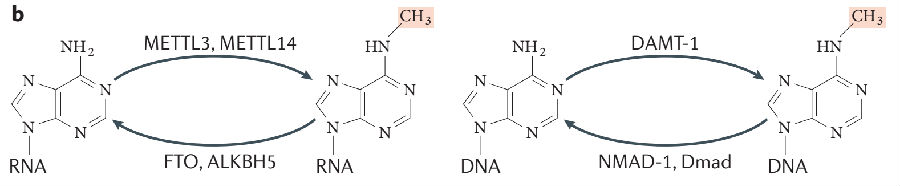
表达丰度的巨大差异,导致RNA m6A是表观研究的大热门,而DNA层面的研究相对较少。
但近几年,DNA 6mA的重要功能逐渐被揭示,其研究前景可谓一片蓝海~
DNA 6mA的研究现状
一直以来,6mA被认为是一种广泛存在于原核生物的甲基化修饰碱基,主要在宿主防御系统中起作用。2015年,Cell杂志连续发表三篇文章,分别在衣藻[3]、秀丽隐杆线虫[4]、果蝇[5]中鉴定了6mA的存在与功能;2016年,Nature发表了小鼠胚胎6mA修饰研究[6]。今年6月,中山大学学者利用三代测序首次获得了中国人DNA 6mA修饰图谱[7]。在顶级期刊的频频露面,使学者们发现6mA在DNA层面的研究并不简单,可能具有重要的调控功能。

DNA 6mA的功能
虽然DNA 6mA表达丰度不及RNA,但近年来多篇文献均表明其在基因转录调控水平起到重要功能,主要包括以下方面:
1. 基因表达调控作用
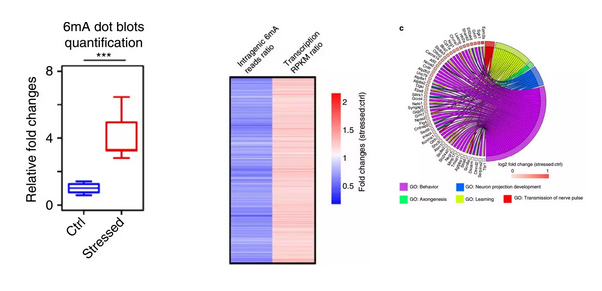
众所周知,DNA 5mC异常甲基化常伴随着相关基因表达的变化。2017年,Bing Yao等[8]研究了环境压力下小鼠脑部的6mA分布变化,发现实验组小鼠脑部6mA修饰显著上调,同时神经系统调节相关基因显著上调,说明在真核生物中,6mA可能行使与5mC类似的功能,通过甲基化状态的改变调节基因表达进而影响生物性状。
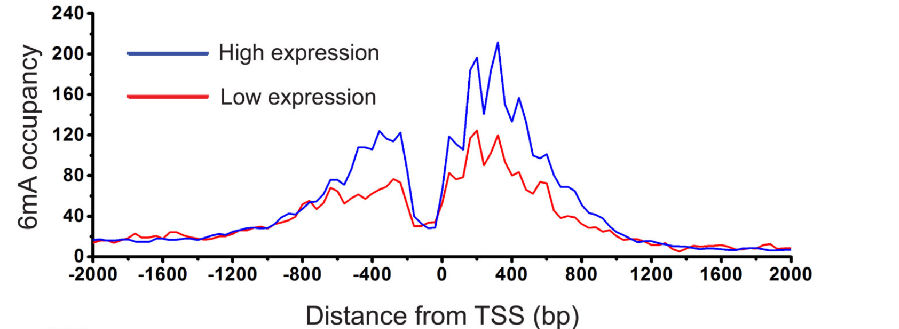
类似的研究在衣藻中也有发现:如图,衣藻中大部分转录活跃基因的TSS附近存在6mA的富集[3]。
2. 参与染色质构象重塑
还是在衣藻的研究中,学者发现6mA通常位于核小体之间的linker DNA上,且linker DNA越长,其上6mA分布越少。目前暂时不清楚是由于linker DNA上的甲基化酶调控6mA的分布,还是6mA对核小体的分布及linker DNA的长度存在调控作用,但6mA确实与染色质构象存在一定关系[3]。

DNA6mA研究手段和高分文章攻略
目前,关于DNA 6mA的研究技术主要有四类[2]:
1. 6mA-IP seq 2. 6mA-RE-seq
3. LC-MS/MS 4. SMRT测序
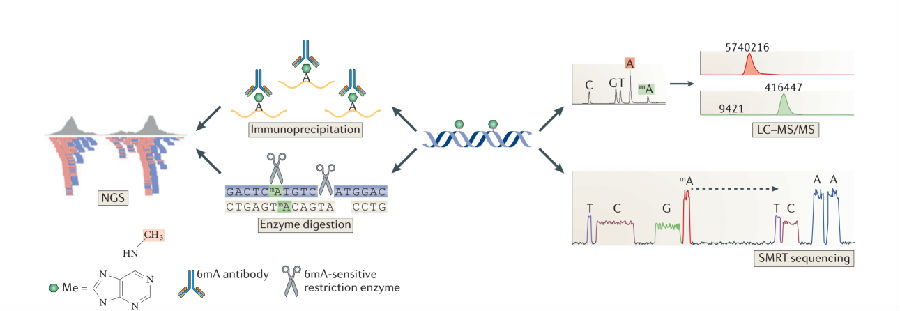
如图,6mA-IP seq与6mA-RE-seq都基于二代测序获得6mA信息,前期分别通过抗体富集和限制性酶切获得含有6mA的片段,性价比高,分辨率相对低;
LC-MS/MS质谱可以定量样本中6mA/A的比例。质谱法的灵敏度很高,可以检测到丰度极低的6mA。但样本污染可能对实验结果造成巨大偏差;
SMRT(Single molecule real-time sequencing)技术基于检测不同甲基化状态碱基的动力学差异进行6mA的鉴定,具有高效、实时等优势,但成本高,且不能很好的分辨6mA和1mA。
综上所述,事实上目前针对6mA的各种研究手段都各有利弊,各位看官还是需要根据自己课题和经费实际情况设计实验方案。
目前,DNA 6mA的高分文章多采取多种手段联合的方法进行研究,如今年7月,华中农业大学周道绣课题组同时使用了6mA-IP seq、LC-MS/MS、SMRT测序对水稻基因组的6mA进行了精确定量和定位[9],成果发表于Nature plants。小编所在的安诺基因团队有幸参与了本文的6mA-IP seq工作,为世界首个水稻基因组6mA图谱的绘制贡献了绵薄之力~


安诺基因团队不仅能提供国内领先的6mA-IP seq技术服务,还建立有完善的表观遗传研究体系。各位老师如果有相关研究课题或者兴趣,欢迎联系我们了解一下相关技术方案,一定能对您的研究有所帮助~
参考文献
[1] Luo, G.-Z. & He, C. DNA N6-methyladenine in metazoans: functional epigenetic mark or bystander? Nature Structural and Molecular Biology 24, 503 (2017).
[2] Luo, G.-Z., Blanco, M. A., Greer, E. L.,He, C. & Shi,Y. DNA N6-methyladenine: a new epigenetic mark in eukaryotes? Nature reviews Molecular cellbiology 16,705 (2015).
[3] Fu, Y. et al. N6-methyldeoxyadenosine marks active transcription startsites in Chlamydomonas. Cell 161, 879-892 (2015).
[4] Greer, E. L. et al. DNA methylation on N6-adenine in C. elegans. Cell 161, 868-878 (2015).
[5] Zhang, G. et al. N6-methyladenine DNA modification in Drosophila. Cell 161, 893-906 (2015).
[6] Wu T P, Wang T, Seetin M G, et al. DNA methylation on N6-adenine in mammalian embryonic stem cells[J]. Nature, 2016.
[7] Xiao Chuan-Le,Zhu Song,He Minghui et al. N-Methyladenine DNA Modification in the Human Genome.[J] .Mol. Cell, 2018, 71(2):306-318.e7.
[8] Yao,B. et al. DNA N6-methyladenine is dynamically regulated in the mouse brain following environmental stress. Nature Communications. 8, 1122 (2017).
[9]Chao Zhou, et, al. Identification and analysis of adenine N6-methylation sites in the rice genome[J]. Nature Plants, 2018.
文章来源于安诺基因

