作为一只生信汪,sam /bam文件是我们最常用的文件,不管你是RNA,DNA,Methy ….还是啥,都要经过sam/bam。今天小编就为大家分享一下,使用samtools工具处理 sam/bam文件的小妙招。
第一步准备文件
1、samtools view -Sx.sam -o xxx.raw.bam (SAM2BAM老司机都知道~)
2、samtools faidx ref.fasta (参考基因组索引)
3、samtools sort xxx.raw.bam xxx.raw.bam.sort
4、samtools index xxx.raw.bam.sort.bam YGQ.raw.bam.sort.bam.bai
然后,show time。
samtools view –X xxx.raw.bam(加入-X参数可以将reads比对信息具象化)。

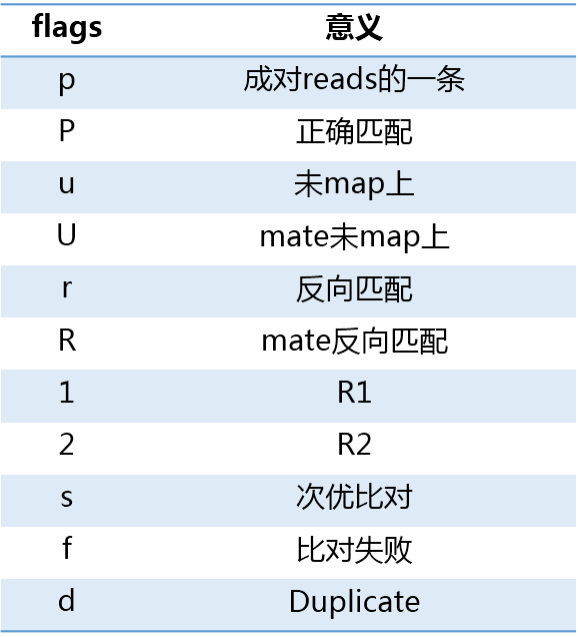
samtools view –F 4 -@ 4 –ubS –t ref.faixxx.sam -o xxx.filter.bam (去除未比对上的序列,四个线程,不对输出进行压缩,输出为bam,输入为sam。 )
[要是您的机器存储紧张,建议您比对时使用管道符“|”来减少消耗。例:bwa mem -t 2 -M –k 30 ref.fasta xxx.1.fq xxx.2.fq|samtools view –F 4 -@ 4 –ubS –t ref.fai -oxxx.filter.bam -]
samtoolsfaidx ref.fasta NC_937428.1:200-4000 (快速提取基因组NC_937428.1染色体上第200到第4000个碱基序列)。
samtoolsdepth xxx.raw.sort.bam > xxx.raw.sor.bam.depth.txt(统计所有位点深度信息)。
samtools depth -q 30 -Q 5 -r NC_937428.1:100-2000 xxx.raw.sort.bam >xxx.raw.sort.bam.depth.txt(统计染色体NC_937428.1上100到2000位点间测序质量大于30,比对质量大于5的点的深度信息)。
samtools flagstat xxx.raw.bam(快速得出比对文件的统计信息)。

从第一行至第十一行分别表示:

samtools tview -d -s -p chr1:170 xxx..raw.sort.bam ref.fasta

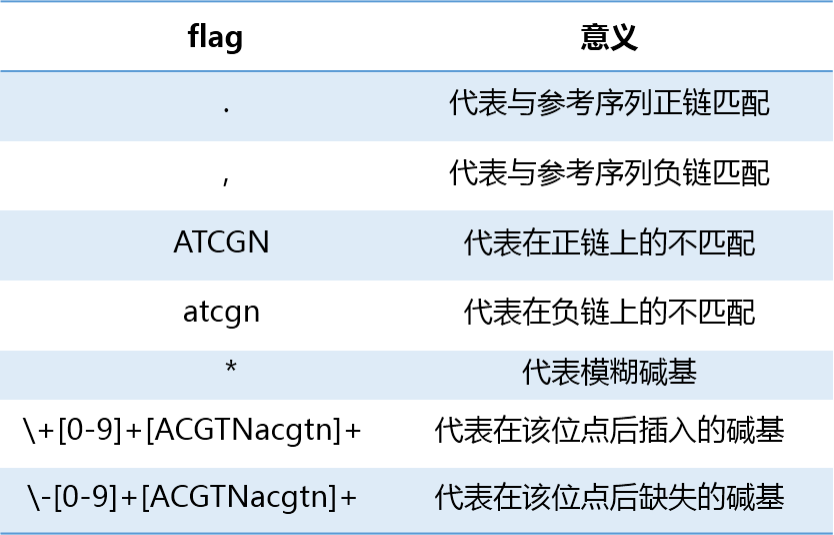

通过tview的各种操作我们可以很直观的看到某些特定位置的比对情况、碱基质量以及深度、覆盖度、变异情况。
通过小编的分享,大家可以发现其实生信分析虽然看起来很难,但是有很多比较简单实用的捷径可以使用。希望今天的分享对大家的日常研究有所助益。也欢迎大家加入安诺基因生信交流QQ群(群号:213357902),这里有专业的生信老司机带你飞~

